Canali
Ultime news

Ultimi articoli
 Le ultime dal Forum |
Correlati TagPassa qui con il mouse e visualizza le istruzioni per utilizzare i tag!
futuro (1)
, archiviazione (1)
.
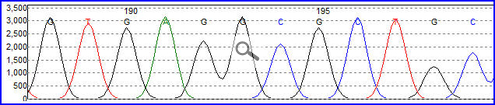
DNA e sistema binario: cosa hanno in comune?Un principio simile è utilizzato dai nostri calcolatori per conservare ed analizzare le informazioni. Come le cellule si esprimono con il linguaggio del DNA (che usa solo 4 "simboli", le basi azotate), così i calcolatori si esprimono con il linguaggio del sistema binario (che usa solo 2 simboli, lo zero e l'uno). Da questo punto di vista, gli "0" e gli "1" rappresentano i nucleotidi dei calcolatori, cioè rappresentano l'unità fondamentale dell'informazione nel mondo dei calcolatori. Immagazzinare informazioni nel DNA è un po' come tradurre da una lingua a un'altra: se è noto l'equivalente binario di un'informazione, ad esempio una parola, tale dato può essere tradotto in nucleotidi fissando delle regole per la "traduzione" (in un senso e nell'altro). A puro scopo dimostrativo, ecco come funzionerebbe il sistema: la parola "MegaLab.it" può essere convertita nel sistema binario con "01001101 01100101 01100111 01100001 01001100 01100001 01100010 00101110 01101001 01110100", e quindi (se A e C = 0, mentre T e G = 1) in nucleotidi con l'ipotetica sequenza "CGAATTCT AGGCATCT AGTCAGTT CGTCCACT CGAATTAC ATGCCACT AGTCAATC ACGATTTA CGTAGCAT ATGTCTAA". In questo caso non è importante che la sequenza abbia "senso" per la conversione del messaggio in aminoacidi, dal momento che l'intento del processo è solo quello di usare la molecola di DNA per conservare informazioni dal significato arbitrario e non necessariamente biologico, e dal momento che nell'esperimento non sono state coinvolte cellule complete e viventi (fattore che potrebbe introdurre indesiderabili variabilità nel messaggio). Come funziona il sistema?I ricercatori hanno "compilato" al computer una sequenza di nucleotidi che traducesse la sequenza binaria di dati destinati ad essere "salvati" nel DNA, avendo dapprima stabilito che i nucleotidi A e C avrebbero codificato per "0", mentre T e G per "1". Il successo dell'esperimento non consiste nell'aver sintetizzato un unico lunghissimo filamento di DNA costituito da una sequenza prestabilita di nucleotidi, ma nell'averlo organizzato in modo tale che possa essere realmente utile dal punto di vista del salvataggio, della ricerca e della lettura dei dati. Il team di Harvard ha suddiviso le informazioni in piccoli frammenti di DNA, ciascuno contenente un numero di nucleotidi codificante per 96 bit (ogni bit è quindi codificato da un nucleotide), a cui è stata aggiunta una sequenza nucleotidica identificativa (una sorta di "etichetta" del frammento) di 19 bit in modo che possa essere collocata in modo univoco nel contesto del messaggio originario completo. Il tutto per un totale di circa 5,27 milioni di "0" ed "1" e quindi circa 54900 piccoli filamenti di DNA. Anche in questo caso, l'analogia con la strategia utilizzata dai comuni dispositivi di archiviazione è evidente: in ogni hard disk è infatti disponibile un numero discreto di "spazi", chiamati "settori", che possono essere occupati ciascuno da una quantità discreta di informazione, ossia di bit. Sintetizzati tutti i frammenti di DNA, il team ha predisposto un sistema simile ad una stampante a getto di inchiostro per fissare ogni frammento di informazione su un supporto, chiamato "microarray chip". In questo modo, ogni frammento viene conservato separato dagli altri, sebbene non mantenendo necessariamente in ordine il messaggio codificato. Il microarray è stato quindi conservato in condizioni controllate (4° C) per circa tre mesi, per poi essere testato. Il sequenziamento delle molecoleIl test consiste chiaramente nella lettura della sequenza dei nucleotidi per ricavare il codice binario dell'informazione conservata. In gergo tecnico, la lettura della sequenza nucleotidica di un filamento di DNA è detta "sequenziamento", ed è un processo che può essere eseguito mediante diverse tecniche. Naturalmente esula dalla scopo di questo articolo illustrare i principi alla base delle più moderne tecniche di sequenziamento, ma è rilevante notare che attualmente queste tecnologie, insieme a quelle che permettono la sintesi chimica del DNA, sono nel pieno di un interessante sviluppo e probabilmente permetteranno nel giro di qualche anno di sequenziare grandi quantità di DNA in tempi contenuti e con costi ancora più bassi di oggi. I ricercatori hanno ripetuto il processo di sequenziamento circa tremila volte su ogni frammento, e i dati tradotti sono stati interpretati e riorganizzati da un computer anche grazie alla presenza del "label" identificativo di 19 bit. Le tecniche di sequenziamento si basano comunemente su reazioni chimiche e sulla rilevazione della fluorescenza emessa da molecole traccianti associate in modo specifico ai diversi nucleotidi, per cui un certo grado di errore è da considerare. Nonostante questo, il team di Harvard - grazie al fatto che le sequenze sono state "lette" più e più volte - ha ridotto gli errori a soli 10 bit su 5,27 milioni. ConclusioniPer via delle intrinseche limitazioni tecniche nelle attuali tecnologie di sintesi e di sequenziamento, non è possibile applicare questi risultati - almeno per il momento - a dati che necessitano di essere aggiornati nel tempo. Attualmente il processo di sintesi della molecola non è in grado di eguagliare neppure lontanamente le velocità raggiunte dalle cellule in vivo, e necessita di tempi abbastanza lunghi. Allo stesso modo, sebbene avanzate, le tecniche di sequenziamento sono solo limitatamente ripetibili, e comunque in genere previa "amplificazione" del materiale da analizzare. D'altra parte potrebbe essere una soluzione molto interessante per conservare quantità spropositate di dati per un periodo indefinito. È nota infatti la stabilità e la resistenza del DNA anche in condizioni non ordinarie: in molti casi è stato possibile sequenziare frammenti di genoma prelevati da mummie o da altri resti anche a distanza di migliaia di anni. Nonostante questi risultati estremi, il DNA tende naturalmente a degradarsi se conservato in condizioni anomale; tuttavia c'è da chiedersi quante centinaia o migliaia di anni potrebbe resistere la molecola se conservata in condizioni controllate! Inoltre, una certa quantità di DNA può contenere facilmente e senza dispendio energetico una mole incredibile di informazioni se rapportate al peso del campione. È stato infatti stimato che un solo grammo di DNA possa contenere le informazioni raccolte in 100 milioni di DVD. Non è da escludere a priori che in un futuro non troppo lontano sia disponibile una tecnologia che permetta di "salvare" e "leggere" rapidamente le molecole di DNA, e magari anche modificarle in punti specifici. Affidare la memoria del genere umano a questa molecola potrebbe avere implicazioni inimmaginabili... oltre rappresentare uno spunto per un nuovo filone di romanzi fantascientifici. Il futuro dell'archiviazione è nel DNA? Segnala ad un amico |
- Unisciti a noi
- | Condizioni d'uso
- | Informativa privacy
- | La Redazione
- | Info
- | Pressroom
- | Contattaci
© Copyright 2025 BlazeMedia srl - P. IVA 14742231005
- Gen. pagina: 1.07 sec.
- | Utenti conn.: 167
- | Revisione 2.0.1
- | Numero query: 43
- | Tempo totale query: 0.27